本站讯 近日,37000cm威尼斯教师柳军涛课题组在生物学国际顶级期刊Cell的子刊Patterns发表了题为“TriNet: A tri-fusion neural network for the prediction of anticancer and antimicrobial peptides”的论文。37000cm威尼斯(威海)为论文第一完成单位,澳国立联合理学院的周婉芸和刘雨菲为论文共同第一作者,柳军涛为论文通讯作者。
抗菌肽和抗癌肽均是由多个氨基酸组成的生物活性肽,抗菌肽能够解决细菌性病原体产生耐药性导致药效降低的问题,抗癌肽有效地控制癌细胞对抗癌类药物的耐药性,进而提高药物的疗效。近年来,包括抗菌肽和抗癌肽已广泛用于各种疾病治疗的临床应用中。因此,准确地识别出抗癌肽和抗菌肽在癌症的治疗和抗菌类药物的设计上发挥着重要作用。抗菌肽和抗癌肽的识别技术主要包括基于生物学实验的方法和基于计算的预测方法。传统的湿实验方法通常昂贵且耗时,因此,开发经济便捷且准确可靠的计算方法显得尤为重要。现有的抗癌肽和抗菌肽预测算法,主要包括特征提取和模型预测两部分,他们大多采用已有的序列特征提取方法和深度学习网络的简单组合来训练模型。尽管这些方法也具有一定的预测性能,但仍存在如下三个局限性:
1.在特征提取方面,没有能够体现肽链全局信息的特征,同时,在理化性质特征提取方面常常直接使用特定的理化性质特征,造成序列特征信息的冗余和低质量。
2.针对不同的特征没有设计与之专门对应的机器学习或者深度学习方法去处理。不少模型都是用一个相同或类似的神经网络去处理多种特征,造成序列特征信息的不合理利用。
3.传统的神经网络模型训练方式都是随机划分训练集和验证集来训练得到最终的模型,无法充分利用现有数据集集合神经网络的偏好性来进行训练和验证集的合理划分。由于数据划分的随机性,导致不同的划分方式在相同测试集下的偏差较大,因此,最终得到的网络模型预测效果极不稳定。
以上这些原因,导致现有的抗癌肽和抗菌肽识别方法识别准确率较低,且泛化能力较差。基于上述考虑,柳军涛课题组联合沙特阿卜杜拉国王科技大学(Kaust)高欣教授课题组提出了一种全新的抗菌肽和抗癌肽的识别模型,并开发出算法TriNet,实现了多肽特征的合理提取、多肽特征的有效利用、以及网络模型的无偏训练,是最终模型的预测效果和泛化能力大幅提升。
该模型TriNet重新定义了多肽的特征提取方法、设计了一个三分支神经网络结构来特异性的处理三类特征、开发出新技术来根据网络的偏好性完成网络的训练,提高抗癌肽和抗菌肽的预测的准确性。1)特征提取模块。本发明首次设计出多肽序列指纹特征,从全局的角度抓取序列的有效信息;本发明重新梳理了多肽序列的理化性质分布信息,解决了信息冗余和低质量问题;最后,再结合多肽序列的进化特征完成最终的特征提取。2)特征处理模块。为了合理的处理上述三类特征,本发明针对它们的各自特点分别设计出如下三种网络模型。设计出一个多通道卷积神经网络接一个通道注意力机制(CNN-CAM)来处理序列指纹信息;设计出一个双向长短记忆网络(Bi-LSTM)来处理序列进化特征;设计出一个多头自注意力网络(Transformer)来处理序列的理化性质分布信息。三个网络分别处理完三类特征后进行一次特征融合,完成抗癌肽和抗菌肽的识别。3)训练模块。本发明首次提出基于网络的偏好性来特异性划分训练集和验证集的新技术TVI,其核心原理为,首先随机划分训练集和验证集,之后,根据网络的偏好性迭代的交换训练集和验证集中的样本来达到最优的划分方式。
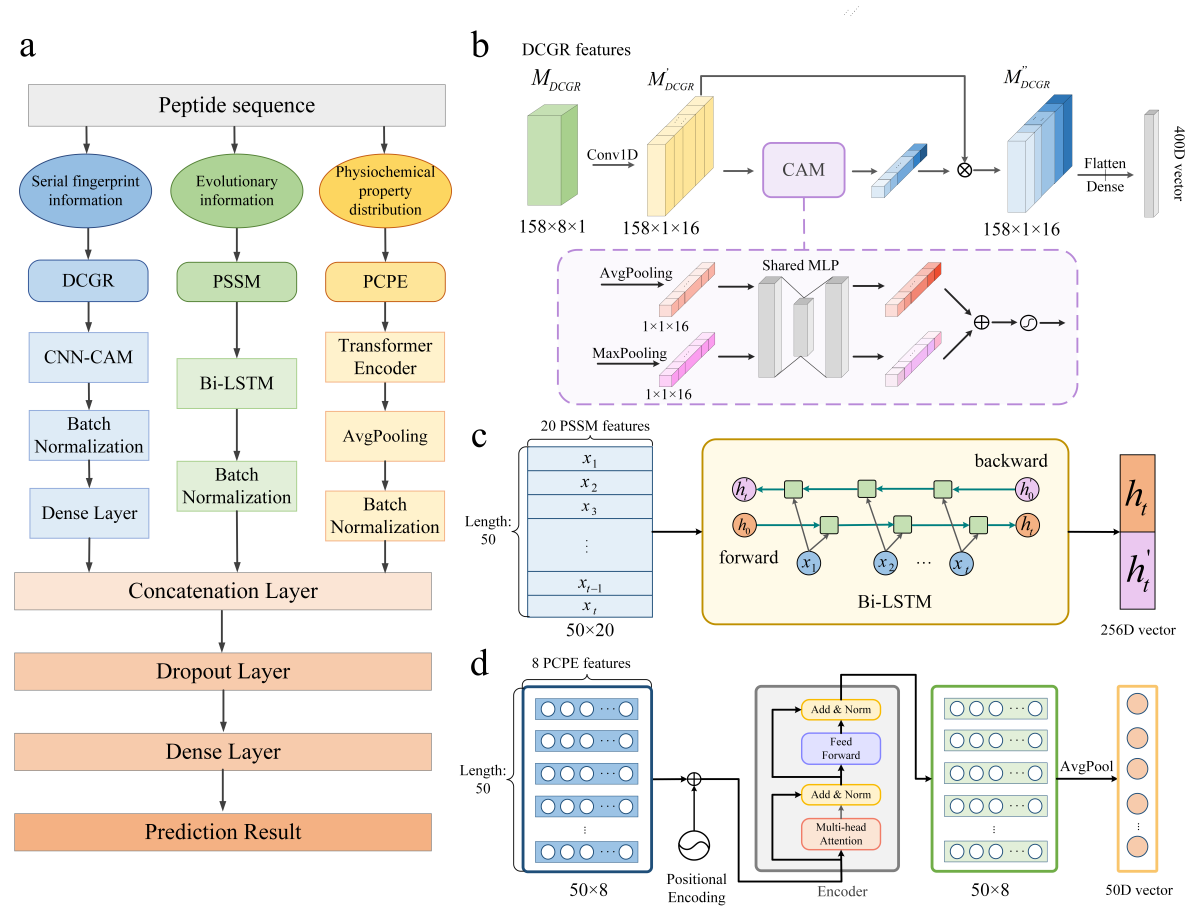
该文章使用了多组最新的数据从特征提取的有效性、无偏训练和传统训练的效果比较、以及与6个著名的抗菌肽预测方法和6个著名的抗癌肽预测方法的预测准确率比较综合验证了本文方法的有效性。比如,本文新开发的无偏训练技术相比于传统的训练方法的准确率(MCC值)提升最高达到16.9%,而且,测试结果显示,本文的无偏训练方法有效降低了神经网络对于数据集划分的偏好性。在进一步地实验中,将其应用在其他多个预测模型中,测试结果显示,应用后达到类似的效果,这充分验证了该无偏训练方法的普适性。另外,在与其他预测方法在所有测试数据集上的多种准确率对比中,TriNet总是达到最高的准确率,且TriNet的准确率(MCC值)提升值最高可达57.2%。总的来说,TriNet无论是在特征提取、网络构建、以及网络训练创新度和实际效果上都表现出明显的优势,该方法有望在癌症治疗药物的研发以及抗菌药物的设计等领域做出一定的贡献,并起到重要的理论指导作用。
文章链接:https://www.cell.com/patterns/fulltext/S2666-3899(23)00039-9
编辑:王祎璠